de UCI Dataset: Lung Cancer
Introducción
Este caso plantea un problema particular, al contarse con muy poca información respecto el contexto y significado de los datos, el amplio numero de atributos y los desbalanceos en los datos de cada atributo. Este ejercicio de clasificación presenta el problema de identificar entre 3 tipos de Cancer de Pulmón a partir de un conjunto reducido de datos pero con muchos atributos.
Resumen y Estadísticas del Set
| Característica | Descripción |
|---|---|
| Cant. de Ejemplos | 32 |
| Cant. de Atributos | 56 |
| Tipo Ejercicio | Clasificación |
| Variable Objetivo | att1: Tipo categórico (1 al 3) |
| Tipos Atributos | Todos los atributos son categóricos, con valores entre 0 y 3 |
Estadísticas
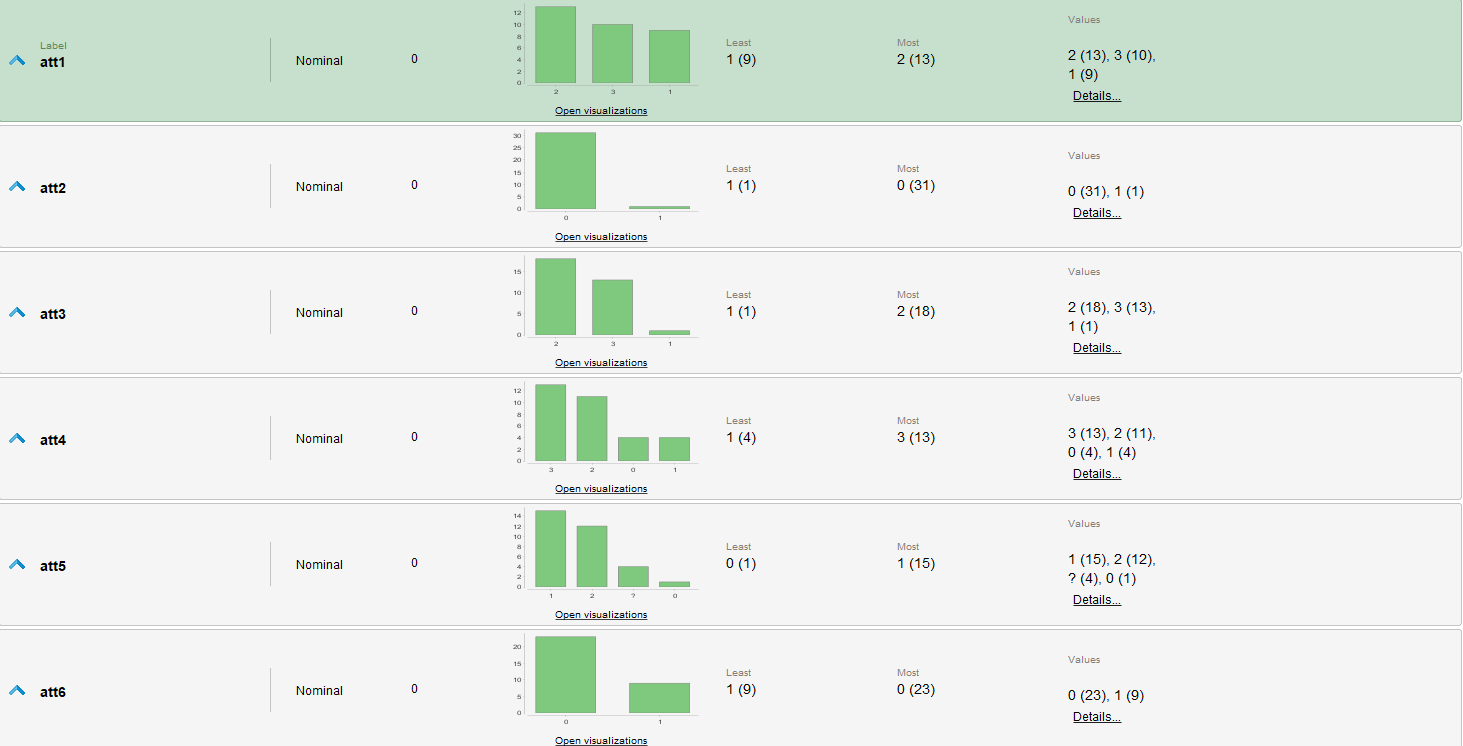
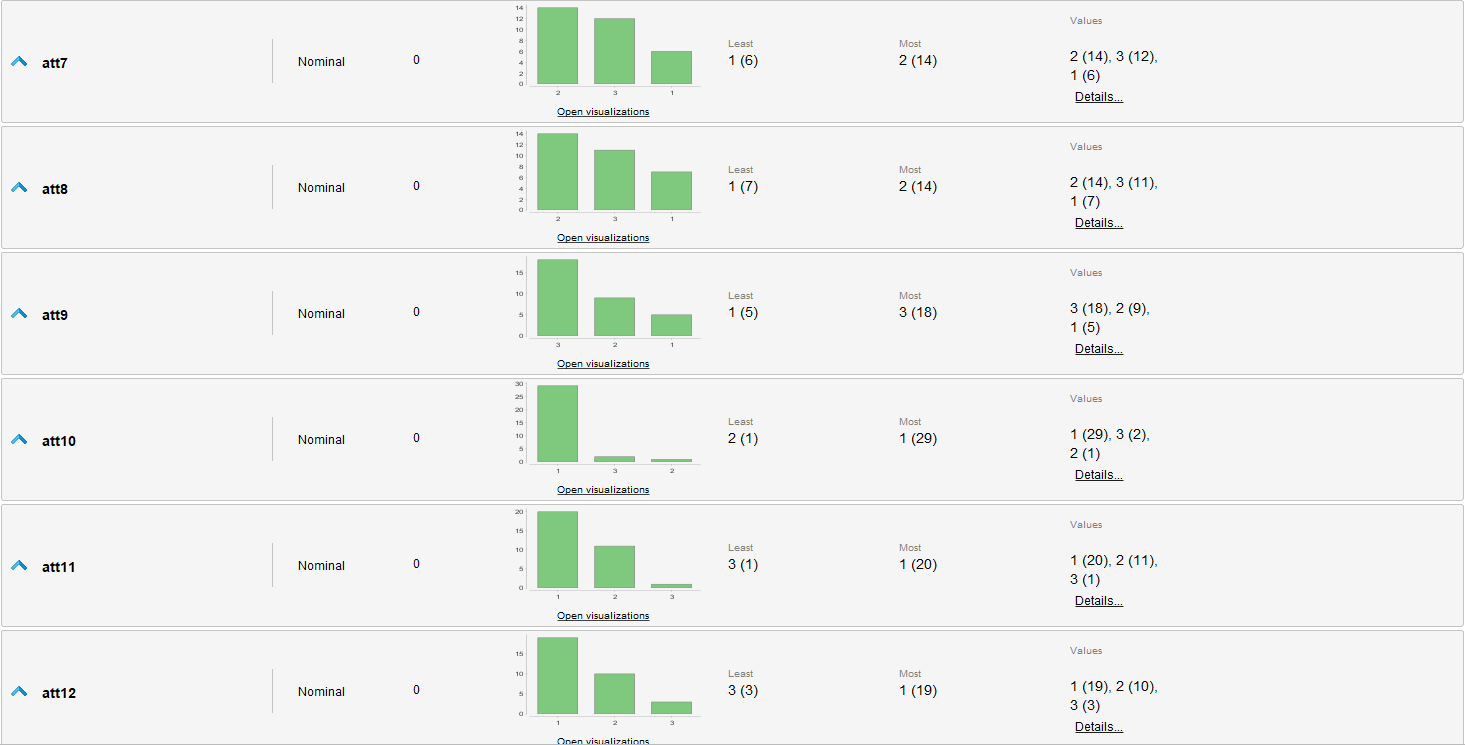





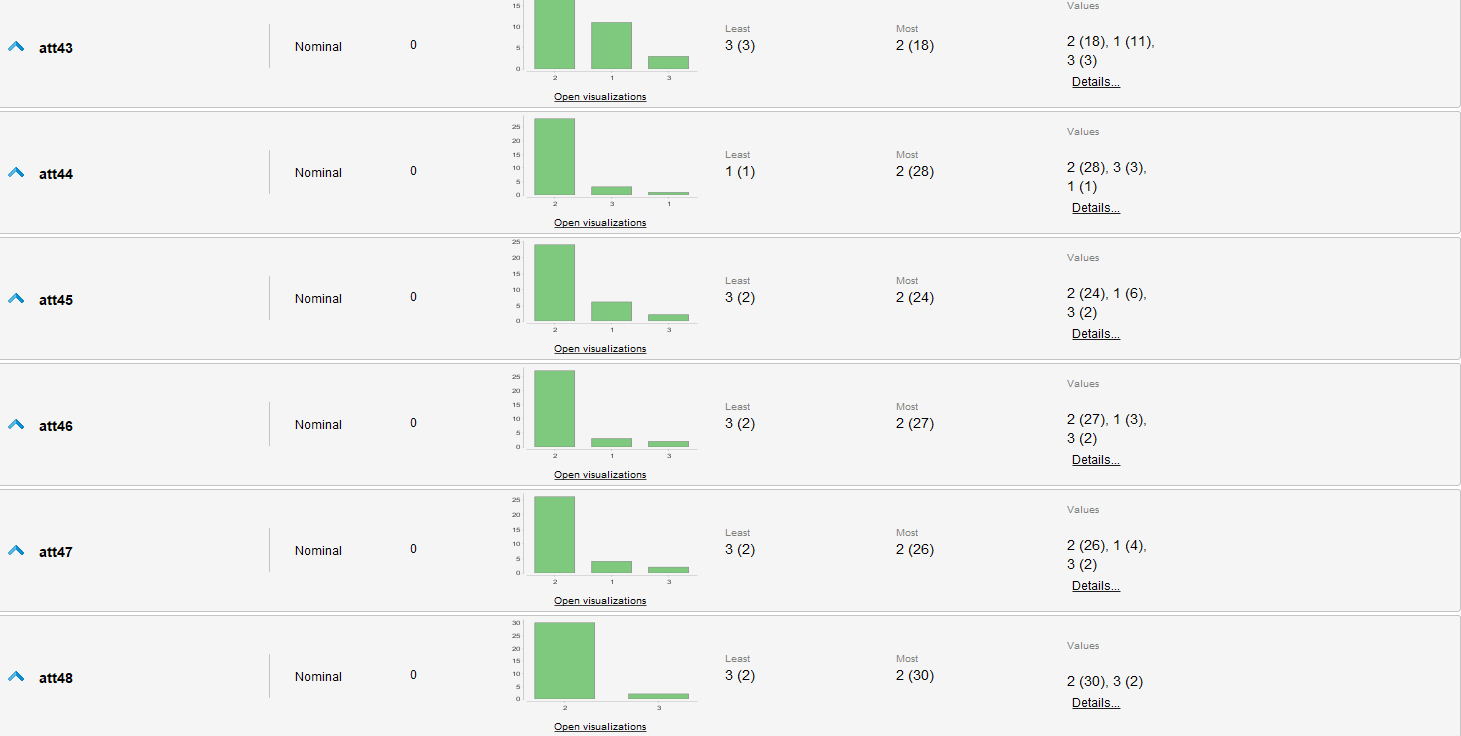

Revisión de Datos
En esta sección elaboraríamos en el significado, implicancias y características de los datos en el contexto del problema, pero para este caso no contamos con la información específica para estos datos.
Resaltamos únicamente que todos los datos son categóricos y aunque tengan valores numéricos no podemos asumir naturaleza ordinal de estos. Similarmente en varios atributos se resaltan cantidades dispares de las diferentes categorías de cada uno.
Procesamiento de Datos
El procesamiento de datos es complejo ya que no tenemos el contexto para discernir de manera racional que atributos aportan o no a la predicción que buscamos hacer, por lo que debemos soportarnos por heurísticas y estadísticas de los datos de por sí. Para el presente caso evaluaremos las diferencias de performance dadas las heurísticas más usadas: Forward Selection, Backward Selection y Evolutionary Selection.
Por otro lado, hay 5 ejemplos con valores faltantes. Normalmente este es un numero insignificante pero dado el reducido numero de ejemplos disponibles decidimos tolerarlos y no eliminarlos. En algunos casos los algoritmos de Feature Selection omiten los atributos que contienen estos valores faltantes (att5 y att39), por lo que el impacto no es muy grave.
Elección de Modelo
Para el presente ejercicio nos planteamos 2 modelos como pretendientes: Naive Bayes y K-NN.
Ambos son buenos algoritmos de clasificación. Elegimos Naive Bayes ya que se especializa en atributos categóricos que en este caso son todos nuestros atributos. K-NN nos permite obtener un modelo performante para el conjunto de datos pequeños, aunque con este arriesgamos introducir la asunción de que nuestras variables categóricas son ordinales, debido al parseo necesario y el calculo de distancia entre puntos. En otros casos la cantidad de atributos sería detrimental al modelo, pero para este caso donde reducimos los atributos a los significativos no es tan fuerte la sobre-dimensionalidad.
Definición de proceso de entrenamiento
Para el entrenamiento utilizaremos Cross-validation de 10 folds, ya que es una bastante estándar de medir el performance de nuestros modelos y debido al poco pre-procesamiento es fácil de implementar.
Por el lado de la Feature Selection, compararemos las heurísticas con los siguientes parámetros:
- Forward Selection: n° máximo de atributos: 30, Criterio de Detención: Sin Mejora.
- Backward Selection: n° máximo de eliminaciones: 10, Criterio de Detención: Sin Mejora.
- Evolutionary Selection: Población: 9, n° máx de generaciones: 30, n° mínimo de atributos: 1.
Procesos
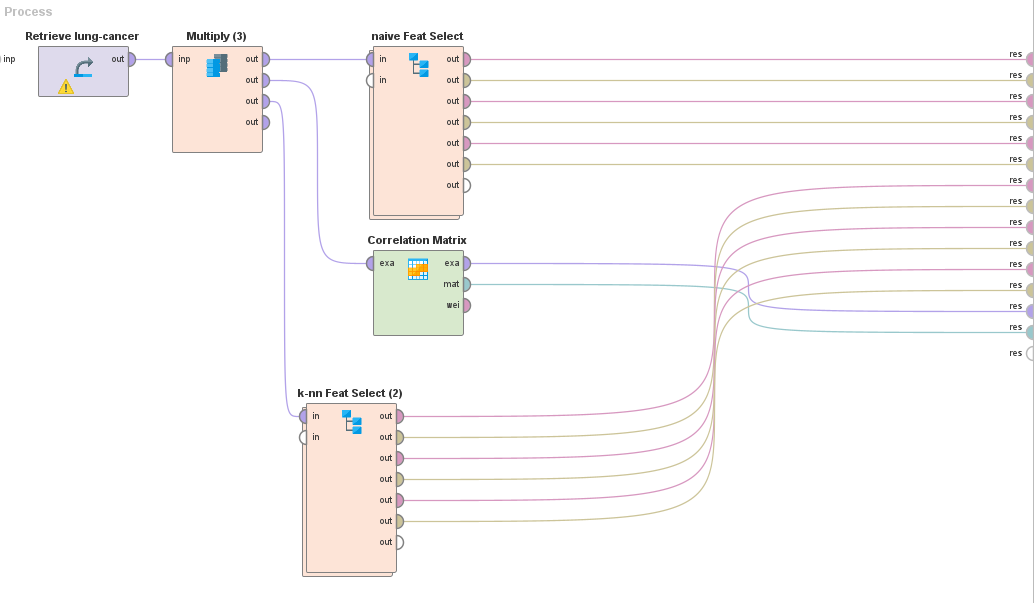
Subproceso "naive Feat Select":
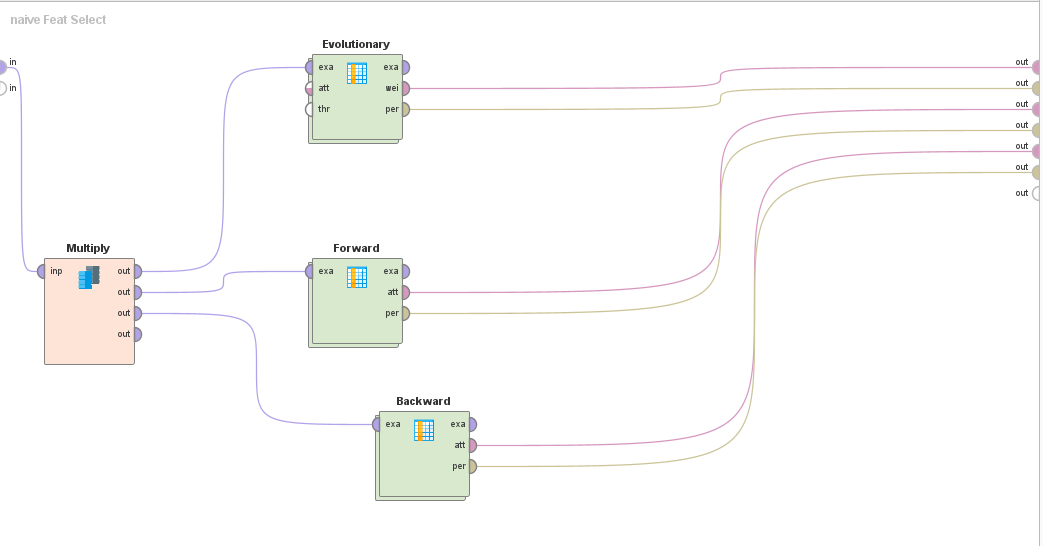
Subproceso "Evolutionary":
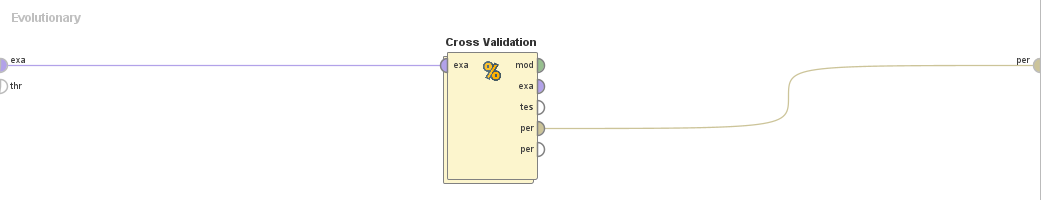
Subproceso "Cross Validation":
Es análogo para las otras heurísticas y modelo, solo se sustituye el bloque correspondiente
Performance
Naive Bayes
Forward Selection
Atributos Seleccionados:
att20, att21, att24, att54
Total: 4
PerformanceVector: - accuracy: 81.67% +/- 25.40% (micro average: 81.25%)
ConfusionMatrix:
| True: 1 | True: 2 | True: 3 | |
|---|---|---|---|
| 1: | 8 | 2 | 0 |
| 2: | 1 | 10 | 2 |
| 3: | 0 | 1 | 8 |
Backward Selection
Atributos Seleccionados:
att2, att3, att5, att7, att8, att9, att11, att12, att13, att14, att15, att17, att18, att19, att20, att21, att22, att24, att25, att26, att27, att28, att29, att30, att31, att32, att33, att34, att36, att37, att38, att39, att40, att41, att42, att43, att44, att45, att46, att47, att48, att49, att51, att52, att53, att54, att55, att57
Total: 48
PerformanceVector:
accuracy: 75.83% +/- 28.45% (micro average: 75.00%)
ConfusionMatrix:
| True: | 1 | 2 | 3 |
|---|---|---|---|
| 1: | 8 | 2 | 0 |
| 2: | 1 | 9 | 3 |
| 3: | 0 | 2 | 7 |
Evolutionary Selection
Atributos Seleccionados:
att2, att3, att6, att7, att9, att11, att14, att17, att19, att20, att22, att24, att26, att29, att30, att32, att34, att36, att37, att39, att42, att44, att47, att48, att49, att53, att54, att55, att57
Total: 29 atributos
PerformanceVector:
accuracy: 84.17% +/- 16.87% (micro average: 84.38%)
ConfusionMatrix:
| True: | 1 | 2 | 3 |
|---|---|---|---|
| 1: | 8 | 1 | 0 |
| 2: | 1 | 11 | 2 |
| 3: | 0 | 1 | 8 |
K-NN
Forward Selection
Atributos Seleccionados:
att7, att41
Total: 2
PerformanceVector:
accuracy: 67.50% +/- 26.77% (micro average: 65.62%)
ConfusionMatrix:
| True: | 1 | 2 | 3 |
|---|---|---|---|
| 1: | 8 | 3 | 1 |
| 2: | 1 | 10 | 6 |
| 3: | 0 | 0 | 3 |
Backward Selection
Atributos Seleccionados:
att3, att4, att5, att7, att8, att9, att10, att11, att13, att14, att16, att17, att18, att19, att20, att21, att22, att23, att24, att25, att26, att27, att28, att29, att30, att31, att32, att33, att34, att35, att36, att37, att38, att39, att40, att41, att42, att43, att44, att45, att46, att47, att48, att49, att50, att51, att52, att53, att54, att55, att56
Total: 51
PerformanceVector:
accuracy: 63.33% +/- 34.29% (micro average: 62.50%)
ConfusionMatrix:
| True: | 1 | 2 | 3 |
|---|---|---|---|
| 1: | 7 | 2 | 1 |
| 2: | 1 | 9 | 5 |
| 3: | 1 | 2 | 4 |
Evolutionary Selection
Atributos Seleccionados:
att2, att3, att6, att8, att9, att11, att14, att17, att19, att20, att24, att30, att33, att34, att35, att36, att38, att40, att42, att46, att53, att54, att55, att56, att57
Total: 25
PerformanceVector:
accuracy: 73.33% +/- 25.09% (micro average: 71.88%)
ConfusionMatrix:
| True: | 1 | 2 | 3 |
|---|---|---|---|
| 1: | 6 | 3 | 0 |
| 2: | 3 | 10 | 3 |
| 3: | 0 | 0 | 7 |
Conclusiones/Observaciones
- Considerando una base de predicción de 33% por elección aleatoria, se lograron buenos modelos con precisiones que superan el 60%
- Se notan en todos los modelos indices sumamente altos de error (+/- 16% hasta +/- 34%).
- Esto puede ser producto de la falta de expresividad y contexto de las variables. Al no poder nosotros trazarles las relaciones correctas dependemos unicamente de los conjuntos optimizados de las heurísticas, los cuales tienen mayor o menor éxito.
- Los Forward Selection tienden a usar muy pocos atributos, mientras que Backward tiende a utilizar todos. Evolutionary suele ser más balanceado.
- El modelo mejor adaptado es Naive Bayes utilizando Evolutionary Selection con una precisión de 84.17% +/- 16.87%
- Este modelo se acopla bien al problema caracterizado por tener todas las variables de tipo categórica, y el método de selección nos permite eliminar el grueso de atributos ruidosos y enfocarnos los más relacionados con la clasificación final.
- El modelo peor adaptado al problema es K-NN con Backward Selection, con una precisión de 63.33% +/- 34.29%
- Esto puede ser dado por la dificultad del calculo de distancias para variables categóricas y debido a que la Backward Selection mantuvo casi todos los atributos, resultando en un modelo que sufre de la Maldición de la Dimensionalidad.